En la entrada introductoria sobre la microbiota os comenté que una de las mayores dificultades a la hora de estudiar esta comunidad es que no puede hacerse por las técnicas clásicas de microbiología. Entre las varias razones que existen, una de las más importantes es que resulta muy complicado hacer crecer las bacterias en el laboratorio. Por lo tanto, y debido a esta gran barrera, en los últimos años se han ido desarrollando una serie de tecnologías que han facilitado en gran medida este proceso, y que consisten en las denominadas “técnicas de secuenciación masiva”. Estas técnicas nos permiten obtener secuencias específicas de los genomas, gracias a las cuales podemos detallar los organismos que conviven con nosotros.
Seguro que algunos de vosotros os habréis puesto a pensar “Espera, espera, ¿sequen… qué? ¿genomas? ¿especificar organismos? ¿pero qué diablos me está contando este tipo, así sin anestesia?”
¡No os preocupéis! El secreto para entender cualquier cosa es dividir el asunto en partes más pequeñas, aprender qué son, y volver a unirlas para volver a tener una visión global del problema. Y es lo que vamos a hacer, así que empecemos.
Genomas y algo de evolución
El primer paso en este camino es saber qué es un genoma, y por qué es importante saber qué estructura tiene. La palabra en sí no es extraña, y hoy en día estoy seguro que la habréis oído en múltiples sitios. Así pues, un genoma es simplemente todo el contenido genético de un organismo. Y cuando nos referimos a todo este contenido genético estamos hablando tanto de los genes como de las secuencias que no son genes.
Ahora os preguntaréis, y bien, ¿qué es un gen? Un gen es la unidad de información de nuestro genoma que es capaz de ser traducido para dar lugar a algo con utilidad. Por ejemplo, gran parte de los genes se traducen a las diferentes proteínas de un organismo, y éstas a su vez ejercen una función específica. Tanto los genes como lo que no es un gen está escrito en el ADN mediante diferentes combinaciones de las famosas cuatro letras A, C, G y T. Es decir, y resumiendo un poco esta parte, un genoma está compuesto por frases con un significado (o utilidad), y que está escrito en este idioma de solo cuatro letras.
Una vez visto qué es un genoma, ahora debemos pensar en lo siguiente. Si recurrimos al clásico ejemplo de que en nuestros genes está escrito una especie de “libro de instrucciones” de nosotros mismos, tenemos que tener en cuenta que, al igual que pasa en un coche o reloj, hay algunas partes que son más importantes que otras. Por ejemplo, si a un coche le quitamos la rueda de repuesto seguirá funcionando con normalidad, pero si le quitamos alguna pieza importante de su motor seguramente deje de hacerlo. Pues bien, en un genoma pasa algo parecido, cuando cambiamos una parte importante lo más seguro es que todo deje de funcionar, o que lo haga mal.
Pensaréis, ¿y a mí qué me importa esto? Pues bien, estoy seguro que os suena la palabra “evolución”. Ésta se basa en el cambio que ocurren en los organismos por la acción de varios factores, como por ejemplo, presiones por el entorno en el que viven. El modo en que un organismo cambia tiene que ver con modificaciones en sus genomas, mediante las llamadas mutaciones, de las cuales se dice, de forma errónea, que son al azar. Por lo tanto, si unimos las ideas del párrafo anterior y la del actual, podemos suponer que, aunque un genoma padezca mutaciones, no todas son igualmente importantes ya que hay genes (o regiones) que son más importantes que otros y que cambiarán mucho menos que otras regiones que no tienen tanta importancia.
Aunque os parezca que nos estamos yendo por las ramas, todo esto es relevante para comprender lo siguiente. Como os he comentado en la introducción, nuestro objetivo es ser capaces de identificar organismos utilizando algún tipo de secuencia. Al decir esto partimos del hecho que cada organismo tiene un genoma diferente a los otros debido a su historia evolutiva. Además, y como seguro que ya inferís con lo pesado que me estoy poniendo, no todas las partes del genoma han cambiado a la misma velocidad a lo largo del tiempo. Habrá partes muy cambiadas y otras no tanto, dependiendo de la importancia de esas partes. Aquí entra en juego la elección de qué gen o región vamos a escoger, y por qué. Pero ya os lo adelanto, el gen que normalmente se escoge para esto es el del ribosoma, y ahora vamos a ver las razones.
El ribosoma
La elección de este actor protagonista no es casual, el ribosoma es una parte bien conocida de la maquinaria celular y ocupa una posición central en la funcionalidad de la célula. Se encarga de la importante y necesaria tarea de traducir la información de los genes a proteínas (los pasos intermedios son ahora irrelevantes). Es decir, y utilizando un ejemplo sencillo, se trata de un señor que coge las instrucciones del mueble de Ikea que acabas de comprar y te lo monta. Ya lo hemos comentado anteriormente, pero proteínas tenemos todos, así que el ribosoma es una parte central en todas y cada una de las células vivas de este planeta. Nada mal, ¿no? Además, la elección del ribosoma tiene cierto carácter histórico, ya que el estudio del mismo nos permitió pasar de los cinco reinos de Whittaker (animales, plantas, hongos, protistas y moneras) a los actuales tres dominios (eucariotas, bacterias y arqueas), aunque esto os lo contaremos en otra entrada.
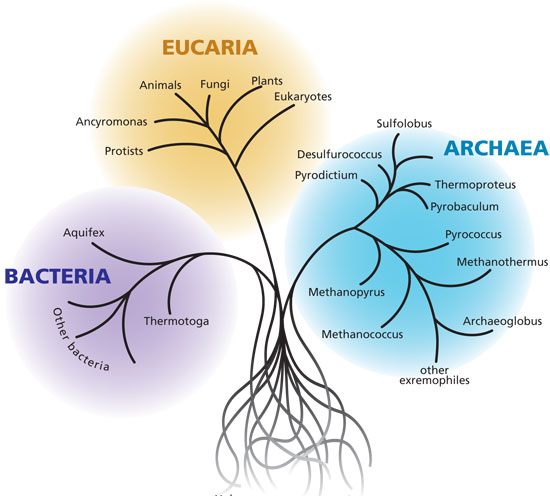
Es lo que se llama un gen marcador, un gen que comparte un gran número de individuos y que además varía lo suficiente (ni mucho ni poco) para poder ver diferencias con cierta calidad. ¡Justo de lo que hemos estado hablando todo este tiempo! Hay un detalle muy curioso en todo esto y el cual nos es de gran utilidad a los biólogos para uno de los pasos más importantes de todo el proceso de secuenciación. Hemos comentado ya que no todos los genes varían con la misma rapidez, pero un detalle sutil de la evolución es que incluso dentro de un gen, no todo varía con la misma velocidad. Extraño, ¿no? La razón de esto es prácticamente lo mismo que antes, pero a pequeña escala: no todas las partes de un gen son igualmente importantes, las hay que son fundamentales (porque, quizás, codifican una parte importante de la proteína) y otras que no lo son tanto y tienen más libertad de variación. Nuestro gen protagonista no podía ser menos, como mostramos en la siguiente imagen. Como podéis ver, hay unas regiones que son muy variables, las cuales nos dan información sobre el organismo; y otras regiones más estables, las cuales son importantes.

La PCR
¿Por qué es relevante este detalle? Pues bien, uno de los pasos de todo el proceso es seleccionar aquello que nos interesa para obtener su secuencia. Ya tenemos a nuestro gen ideal y ahora solo nos falta la forma de poder conseguirlo. Y si además podemos conseguir una buena cantidad del mismo, pues mejor que mejor. Pues eso es relativamente sencillo debido a uno de los inventos más importantes de las últimas décadas para los biólogos en general, la PCR. Las siglas de esta extraña cosa significan “reacción en cadena de la polimerasa”. La invención de esta tecnología hizo que le otorgaran un Nobel a su excéntrico creador, Kary Mullis, el cual merece un par de entradas para él solito.
Pero no nos vayamos por las ramas, ¿cómo funciona la PCR? La polimerasa, esa cosa que aparece en el nombre, es una de las proteínas fundamentales de todo ser vivo, y su función es copiar cadenas de ADN en otras nuevas, algo muy útil si quieres que tu descendencia siga teniendo información genética. Pero claro, a nosotros no nos interesa copiar ADN así, en general, sino que queremos copiar una parte muy específica: el gen del ribosoma. Realmente, para que la polimerasa empiece a funcionar, se le ha de dar un pequeño trozo que inicia toda la reacción, una especie de cebador. Este cebador es una pequeña secuencia que se une a nuestra cadena de ADN (y por lo tanto se tiene que parecer algo a ésta) y que le permite a la polimerasa darse cuenta de que tiene que empezar a copiar. En una PCR uno pone dos tipos de cebadores, uno por cada uno de los extremos del trozo que quiere copiar, y lo que ocurre en esta tecnología es que, utilizando la función de copiar, uno va repitiendo el proceso de forma cíclica. Daos cuenta, en el primer ciclo habremos copiado la información una vez y tendremos dos copias, en el segundo ciclo tendremos cuatro copias, en el tercer ciclo ocho copias… Si lo repetimos un número de veces, pongamos entre 20 y 30, tendremos ¡miles de copias! Todo el proceso está resumido en la siguiente imagen.

Pues bien, volviendo al tema que nos interesa, lo que nosotros hacemos para elegir el gen del ribosoma realmente no es elegirlo entero sino solo alguna o algunas de sus partes más variables. Para hacer esto elaboramos unos cebadores que se pegan en sus partes más conservadas, que si recordáis, rodean a las partes variables. Estas partes más conservadas tienen la gran ventaja de que no han variado casi nada entre bacterias, por lo que uno puede construir unos cebadores “universales” que se pegan a las secuencias de los ribosomas de todas las bacterias de forma indiferente. Vaya, no me digáis que no es una solución inteligente, ¿eh?
¡Pues ya está prácticamente hecho! Recapitulando, queríamos estudiar quién había en nuestra población bacteriana de interés. Para ello hemos escogido un gen marcador porque varía entre organismos, pero tampoco mucho para que no nos volvamos locos. Lo hemos amplificado a lo bestia con nuestra flamante técnica de la PCR, y ahora solo queda meterlo en el secuenciador. Un secuenciador es una máquina que, mediante diversos métodos basados en las propiedades del ADN, nos permite saber qué letras hay en cada una de las secuencias que le metemos. Tras esto, obtendremos un buen número de secuencias, las cuales asignaremos a organismos mediante programas bioinformáticos, que no es nada más que técnicas informáticas aplicadas a problemas biológicos.
¿Queréis saber cómo funciona un secuenciador, o cómo se asignan las secuencias a organismo? ¡Pues entonces seguid conectados a ULUM en las próximas semanas!
Daniel Martínez Martínez (@dan_martimarti) es licenciado en Ciencias Biológicas por la Universidad de Valencia, donde también realizó el máster Biología molecular, celular y genética. Realizó su doctorado a caballo entre el FISABIO (Fundación para el fomento de la investigación Sanitaria y Biomédica) y el IFIC (Instituto de Física Corpuscular). Su labor investigadora está centrada en el estudio de la relación entre la composición funcional y de diversidad de la microbiota humana, y el estado de salud-enfermedad de los individuos. Durante los últimos años ha mantenido una actividad de divulgación científica escrita, además de participar en la organización de eventos como Expociencia. Actualmente trabaja en el Imperial College de Londres.

Censando a la microbiota comentarios en «2»